1970년대 쯤 "유전공학(genetic engineering)"이라는 분야가 등장했다. 요즘에도 쓰이지만 이 말은 "유전자 재조합 기술(recombinant DNA technology)"를 의미한다. 이러한 재조합 분자를 생성하기 위하여 클론 될 DNA뿐만 아니라, 벡터도 통제된 방법으로 특정한 지점에서 절단된 후 함께 연결되어야 한다.
절단과 연결은 최근 몇 년 동안 다양하게 발달해 온 DNA조작 기술의 두 가지 예이다. 절단과 연결뿐만 아니라 DNA분자를 짧게 혹은 길게 할 수 있으며 RNA나 새로운 DNA 분자로 사본할 수 있고, 특별한 화학기능기를 첨가 혹은 제거함으로써 수정할 수도 있다. 시험관 속에서 수행할 수 있는 이러한 모든 조작들은 유전자 클로닝뿐만 아니라 DNA생화학, 유전자 구조, 유전자 발현의 조절등과 같은 기본 연구에 기초를 제공한다.

세포에서 DNA ligase는 DNA 복제동안에 이중가닥 DNA 분자 내에서 일어나는 한 가닥 파손(불연속)을 복구한다. 대부분의 유기체의 DNA ligase는 두 개의 각 이중가닥 DNA 단편을 결합시킬 것이다. 이 반응을 촉매화 하는 효소를 DNA ligase라고 한다.
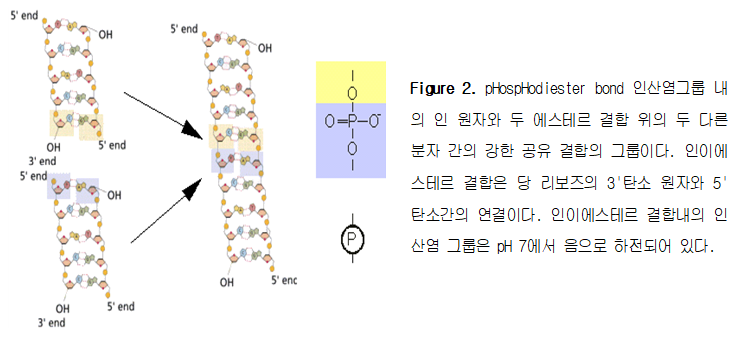
DNA ligase의 활동방식 살아있는 모든 세포가 DNA ligase를 생성하지만, 유전공학에 사용되는 효소는 파아지에 의해 감염된 대장균(E. coli) 박테리아에서 정제된다. 세포내에서 이 효소는 이중가닥 분자의 한 가닥에서 일어날 수 있는 불연속을 복원하는 중요한 역할을 한다. 불연속은 인접하는 뉴클레오티드(nucleotide) 사이에 인산이에스테르(pHospHodiester) 결합이 없는 바로 그 지점이다(이와 반대로 ‘절단’(nick)은 하나 혹은 그 이상의 뉴클레오티드가 없는 곳이다).
불연속은 세포의 DNA 분자 파손을 우연히 야기시킬 수 있지만, 그것은 DNA 복제와 재조합과 같은 과정의 자연적인 결과이다. 그럼으로, ligase는 세포내에서 몇 가지 중요한 역할을 한다. 시험관에서 정제된 DNA ligase는 단일가닥 불연속을 복원시킬 뿐만 아니라 다른 두 개의 DNA 분자를 결합시키거나 같은 분자의 양 끝을 결합시키기도 한다. 두 분자를 봉합하는 화학반응은 각 가닥에 하나씩 두 인산이에스테르결합을 형성하는 것을 제외하면 불연속의 복원과 꼭 같다.
sticky말단은 ligation효율을 증가시킨다. 두 개의 blunt말단 단편이 결합하는 반응은 시험관속에서 일어날 수 있지만, 아주 효율적이지 않다. 왜냐하면, ligase는 ligation되는 분자를 ‘붙잡아’ 둘 수 없어서 양 끝이 우연히 만나도록 기다려야 하기 때문이다. 가능하다면 분자의 양끝이 올바르게 만날 기회를 증가시키기 위해서 고농도 DNA를 사용하여 blunt말단 ligation을 시켜야 한다. 이에 반해, 상보적인 sticky말단의 ligation은 훨씬 효과적이다.
왜냐하면 효소가 작용할 수 있도록 비교적 안정한 구조를 형성하는 수소결합에 의해 상보 sticky말단이 염기쌍을 이룰 수 있기 때문이다. 만약 인산이에스테르결합이 아주 빨리 형성되지 않으면 sticky말단이 다시 떨어져 나갈 것이다. 그렇지만, 이러한 일시적인 염기쌍 구조는 두 말단이 서로 접촉할 시간을 늘려 줌으로 ligation의 효율을 증가시킨다.
실험 방법
1. 실험 재료
준비된 vector와 insert는 다음과 같다[Table1]. quantification위해 1× TAE buffer, 6× dye, EtBr, agarose gel powder, 1kb Marker를 사용하여 전기영동한다. 도구로는 UV illuminator, 1.5㎖ e-tube, centrifuge, micropipette, tip을 사용하였다. 정량한 후 ligation buffer, T4 ligase, 10×T4 ligase reaction buffer[Table.2], D.W.에 vector와 insert를 넣어 ligation mixture를 만들었다.
2. Electrophoresis
준비해 놓은 vector와 insert를 정량하기 위해 1% agarose gel(1% TAE buffer 35㎖ + agarose 0.35g)에 1㎕ loading dye와 3㎕ sample을 섞어 loading 한 뒤 100V로 30min간 running하여 UV를 조사해 band를 확인한다.
3. Quantification
전기영동을 이용한 quantification은 1% agarose gel(1× TAE buffer 35㎖ + agarose 0.35g + 20000× EtBr 1.75㎕)을 만들어 sample과 6× loading dye를 섞어 well에 loading 한 뒤 100V로 running하여 UV illuminator로 band를 확인한다. band의 두께와 진하기를 비교하여 vector의 mole비율을 설정하고, vector 농도에 따른 insert의 농도를 계산하여 reaction volume을 정한다(보통 vector의 mole비율과 insert의 양은 1:3).
4. Ligation
reaction volume에 맞추어 reaction을 design한 후 ligation buffer, T4 ligase, 10×T4 ligase reaction buffer, D.W.에 vector와 insert를 넣어 ligation mixture만든다. ligation mixture는 16℃에서 overnight한다.
[분자유전학실험]DNA ligation 레포트
1. 실험 이론 및 원리 가. 실험 요약 gene cloning의 1단계는 우리가 원하는 유전자를 얻어내는 것이었다(DNA extraction). DNA ligation은 gene cloning의 2단계인 우리가 원하는 유전자와 vector를 결합시키는 것이다. 이 과정은 gene cloning의 마지막 단계인 transformation을 하기 위해서는 꼭 필요한 과정이다. 우리는 ligation을 하기 위해서 준비해 놓은 vector와 insert DN
www.happycampus.com


댓글